Una vez dada la existencia del TTSS, el paso siguiente
sería la adición de un filamento. Matzke y muchos otros sostienen que
filamentos simples a base de proteínas son fáciles de hacer —dando como ejemplo
la polimerización de la hemoglobina en pacientes con anemia drepanocítica, que
es el resultado de una única mutación puntual. Este modo de pensar subestima
los requisitos más específicos que se necesitan para formar un filamento funcional de cualquier tipo.
Por ejemplo, las partes de un filamento aleatorizado,
como las que forman la “hoz” de hemoglobina, son muy propensas a agregarse en
grumos o hebras largamente enredadas antes de que sean transportadas a través
de cualquier tipo de poro a la superficie exterior de la célula. Obviamente eso
no sería útil. Además, incluso si tales monómeros de filamento se hacen llegar
a la superficie exterior sin que ocurra una aglutinación, estos tienen que
adherirse preferentemente al lugar correcto. Eso requiere de características
vinculantes bastantes específicas. ¿Cuáles son las probabilidades de que un
monómero de filamento al azar adquiera también tales características de unión?
Estas posibilidades se traducen en una enorme cantidad de tiempo —en promedio.
Hay un montón de otros problemas potenciales para
producir y organizar monómeros de filamentos. ¿Qué sobre la degradación? ¿Qué sobre
el transporte a través del canal y la selectividad del mismo? ¿Qué sobre
pegarse a la parte interior del canal y la obstruir la vía? ¿Qué pasa si el
filamento terminó formando un núcleo sólido en lugar de un núcleo hueco? ¿Cómo
harían el resto de los monómeros de filamento para adherirse uno a uno en el
extremo del mismo? ¿Qué si el extremo no se encuentra controlado y tapado por
un tipo diferente de proteína que coloca cada pieza nueva de proteína en el
lugar correcto? ¿Cuáles son las probabilidades de que cualquiera de los filamentos
originados vía evolución que se adhiera a la maquinaria de exportación genere
un beneficio en un ambiente determinado, incluso aunque fuese un
"simple" filamento de anclaje?
No sólo que las partes cada vez más y más específicas del
filamento tienen que pegarse entre sí como con el aparato de secreción de la manera
correcta, sino que deben formar un filamento cuyo extremo distal es capaz de
pegarse a otra cosa que no sea en sí la propia superficie de la bacteria. Por
encima de todo lo demás, eso puede ser poco difícil de conseguir. ¿Cuáles son
las probabilidades de que un gen capaz de codificar estas proteínas
especializadas de filamento simplemente surja para que luego su producto sea
secretado de forma específica por un poro de transporte activo ya existente?
Con fin de dar una respuesta a esta pregunta, primero vamos
a considerar lo que se necesita para producir incluso el filamento bacteriano
funcionalmente más “simple” —el pilus P.
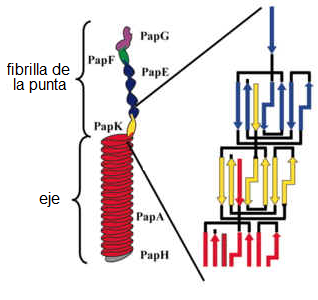
El pilus P funciona como un ancla de unión entre las células
bacterianas y otras células. Se trata de un filamento hueco y delgado que
adelgaza más cerca del extremo. En ese lugar hay una proteína que se une
específicamente a ciertos tipos de moléculas de azúcar en ciertos tipos de células
(como las células del riñón). A pesar de que este pilus es en realidad tan simple
como aparenta y que su función parece ser bastante trivial, se encuentra
codificado por 10 o 11 genes —esa es también la cantidad aproximada de genes
que codifican para el complejo del sistema de secreción de tipo III (TTSS). La
porción proximal gruesa está formada por proteínas PapA, la porción distal más
delgada por proteínas PapE, y el extremo por PapG (la "adhesina"
específica que se une a los azúcares). También hay un adaptador, PapF, que une
a PapG y PapE, y otro adaptador, PapK, que une a PapE con PapA. [14] En total, 5 proteínas diferentes
que se unen en un orden muy específico. ¿Cómo se logra ese orden?
Bien, esto se realiza a través de una complicada
interacción con proteínas "chaperonas". Pero primero, la célula tiene
que construir una vía de exportación multiproteica denominada la ruta Sec, para
porder volcar el material del citoplasma en el espacio periplasmico. El desafío
para aquellas bacterias gram-negativas "que quieren" hacer crecer un
pilus es conseguir que el filamento logre penetrar la membrana externa. Esto
requiere de una cierta coordinación fantástica. Primero, todas las subunidades
del pilus se exportan en un estado no plegado hacia el periplasma a través de
la ruta Sec para luego plegarse. Sin embargo, si esto se deja sin ningún
control, ellas formarían acumulaciones desorganizadas. Por lo tanto, se
requiere de una proteína chaperona, PapD, para evitar este problema y para
ayudar en el pliegue de estas proteínas para que adquieran la conformación correcta,
y lo realiza a través del mecanismo DSC (donor strand complementation). Las
partes del filamento, por sí mismas, son muy inestables y nunca se pliegan
correctamente. Y la proteína PapD no tiene ninguna otra función conocida.
Lo que ocurre después es que el complejo subunidad-chaperona
interactúa con un canal proteico en la
membrana externa, conocido como PapC. Este canal es lo suficientemente grande
como para que la extremidad distal del filamento pueda pasar, pero no la parte
proximal. PapD, la chaperona, le cede la subunidad de pilus a la PapC, que a su
vez ayuda en la unión de esta con el filamento en crecimiento donde cada
subunidad contribuye con una cadena a fin de estabilizar y completar el pliegue
de su vecina.
Por supuesto, existen numerosas subunidades estructurales
que participan en la funcionalidad del pilus P. La fimbria P (o pili P) tiene
mayor cantidad de subunidades estructurales en la bacteria E. coli (con 9 subunidades estructurales). Sin embargo, las fimbrias
del tipo 1 se construyen a partir de 7 subunidades, y las fimbrias YQI,
recientemente caracterizadas, muestran una organización genética en la que
participan solo 4 subunidades estructurales [19]. Tenga en cuenta también que la E. coli presenta numerosos operones fimbriales con un número
variable de subunidades estructurales. Sin embargo, "todas las adhesinas
fimbriales comparten una organización genética común, en la que los genes
reguladores de adhesina preceden al gen de la subunidad mayor, que es seguido
por la chaperona periplásmica, una estructura ordenadora (“ujier”) en la
membrana externa, y por último los genes de adhesina. Esta organización del
grupo de genes se ve en las fimbrias del tipo 1 (fim), fimbrias S (sfa),
fimbrias F1C (foc) y las fimbrias reconocedoras
del antígeno Dr (dra). La organización
del pack de genes de la adhesina yqi
difiere en el hecho de que las posiciones de la proteína “ujier” y chaperona se
encuentran invertidas, lo que también ocurre en el caso del grupo de genes de
la adhesina pap de las fimbrias P,
aunque el motivo de esto, sea funcional o biológico, todavía no se ha
aclarado". [19]
En pocas palabras, todas las fimbrias o pili bacterianos
comparten los mismos elementos indispensables y misma programación estructural
indispensable —que es bastante compleja, incluso en los casos más simples que
se conozcan de fimbrias funcionales.
Así, incluso algo tan relativamente "simple"
como la construcción de un pilus parece bastante complicado en comparación con
el paso evolutivo que se ha propuesto en la evolución del filamento. Es muy
difícil producir un filamento "útil" —al menos eso es lo que parece.
Pero, digamos que un filamento como ese pudiera evolucionar de alguna manera.
¿Cómo va a evolucionar hacia un flagelo? Un flagelo tiene que ser capaz de
secretar proteínas a medida que está siendo construido. El problema es que no se
ha demostrado que algún pilus P pueda secretar proteínas —quizás debido al
tamaño pequeño del canal o la falta de una fuente de energía para el bombeo de
proteínas hacia el exterior. En cualquier caso, un pili como ese incluso sería
muy diferente a un flagelo en un aspecto muy importante. El pili se construye
de arriba hacia abajo, donde cada nuevo monómero que se agrega empuja hacia
arriba y hacia afuera al pilus existente. Los flagelos, por el contrario, se construyen
de abajo hacia arriba; se añade cada nuevo monómero al extremo distal o punta a
medida que va creciendo hacia el exterior sobre el flagelo existente (vea la siguiente animación de la biosintesis del pili y comparela con la animación del ensamblaje flagelar provista en el post anterior).
El fallecido Robert Macnab, ex-profesor de biofísica
molecular y bioquímica de la Universidad de Yale, quien también estudió a los flagelos,
señaló que el mecanismo de construcción del flagelo es "un proceso mucho más
sofisticado que cualquier otro que pudiéramos haber imaginado alguna vez" [8] Mas tarde llegaría a admitir:
"imaginamos que no sería posible que el sistema funcione si tuviese una
complejidad mucho menor." [9]
El filamento
flagelar
Entre las bacterias existe un género interesante que
carece de motricidad, conocido como Shigella;
tiene genes flagelares, pero no sintetiza ningún flagelo. Algunas cepas de
Shigella tienen menos de estos genes que otras cepas, pero en ciertas cepas, el
único gen que falta es el gen FliD. El gen FliD codifica para la proteína cap
que se coloca en la punta del filamento. Sin tal proteína de cubierta denominada
FliD, los monómeros de flagelina (FliC) que forman el filamento se pierden.
Además de eso, sin la FliD, las partes FliC simplemente no se ensamblarán
correctamente (ver animaciones de Keiichi Namba et . Al. [12]). Esta
proteína cap luce como un anillo pentagonal que se inserta encima del filamento
flagelar hueco. Cada una de las 5 partes del pentámero FliD tiene un apéndice a
modo de pata que apunta hacia abajo y se relaciona estrechamente con los
monómeros de filamento. Sin embargo, existe una ligera falta de coincidencia.
Esta cubierta tiene 5 patas, mientras que el extremo del filamento presenta 5,5
subunidades de flagelina en su circunferencia. Por lo tanto, siempre queda un
poco de grieta en un punto entre la cubierta y el filamento. La siguiente
subunidad se agrega justo en ese punto del flagelo en crecimiento. A medida que
la subunidad va pasando por el lugar abierto, la cubierta gira de manera que un
nuevo punto se abre al lado del que se acaba de llenar. Así que, como la
cubierta da vueltas y vueltas, a 10 revoluciones por segundo, nuevos monómeros
de flagelina (FliC) se añaden uno a uno, en total, 50 por segundo [8].
Lo más interesante de todo esto es que los extremos de
las subunidades de flagelina se encuentran en un estado no-plegado —así es como
viajaron a través del conducto del filamento hueco. Una de las razones de esto
es que la flagelina plegada logra cierta torsión en el medio que hace que sea
demasiado grande como para viajar a través del tubo. Por sí mismas, las
subunidades de flagelina no pueden plegarse correctamente. Por lo tanto, se
requiere la cubierta FliD tanto para plegar como para colocar los monómeros de
flagelina. Es otras palabras, es un tipo de chaperona. Además, la zona hueca
justo debajo de la cubierta es de aproximadamente el doble del tamaño que el
resto del tubo y lo suficientemente grande como para permitir el plegado de una
subunidad monomérica. El giro de la tapa combinado con interacciones proteína-proteína
favorables proporciona la energía para este proceso de plegado —ya que no hay
ATP involucrado. [8]
En breve, sin esta cubierta altamente especializada, las
unidades de flagelina no pueden en absoluto auto-ensamblarse para formar un
filamento tan ordenado. Y ni la proteína cap ni los monómeros de flagelina
tienen cualquier otra función celular. ¿Cómo es que la proteína cap se coloca
en la posición correcta en el extremo del filamento y como es que otras
proteínas cap ya no son enviadas por el tubo una vez hecho esto? Una vez más,
se requiere una chaperona específica para el montaje de la cubierta y la prevención
de la agregación prematura.
Para contrarrestar este argumento, se hace la aseveración
de que debido a que las unidades de proteína tubular flagelar, FliL y FliK, no
necesitan una cubierta para su montaje adecuado, la adición de una cubierta
para este sistema consistió en una modificación evolutiva tardía con el fin de
mejorar la velocidad y la eficiencia. [1]
Un problema potencial para este argumento es que FliL y FliK son solo proteínas
de unión. Su función es unir parte del gancho del flagelo (FlgE) con el resto
del flagelo (FliC). Por sí mismas, no forman la estructura flagelar. Incluso si
lo hicieran, esto no explicaría cómo las subunidades de flagelina (FliC) podrían
auto-ensamblarse sin una proteína cap o cómo podrían haber evolucionado sin la
co-evolución de tal cubierta, que es muy específica —requiriéndose un gran
número de cambios de residuos altamente especificados para la obtención de la
mínima ventaja selectiva.
Pero ¿qué sucede con la hipótesis de la "primera
cubierta" en la que la proteína cubierta evoluciona, debido a sus
propiedades adhesivas, y es mejorada por posterior evolución de las proteínas
del pilus que la terminan llevando hacia el exterior de la célula? De nuevo, ¿en
cuánto tiempo podría un monómero de proteína flagelar llegar a ser lo
suficientemente específico como para interactuar con una cubierta, y de una
manera tan compleja?
Las explicaciones de Matzke no consiguen ser más
detalladas que lo que hemos venido viendo. Si estos pasos evolutivos eran tan
fáciles de atravesar, podrían ser fácilmente probados en el laboratorio. Sólo
se tendría que eliminar en una bacteria el gen FliC que codifica para la flagelina
y observar si sus descendientes van a evolucionar de nuevo un flagelo con la
cubierta “preestablecida”. Hasta donde sé, no ha habido experimentos como este
que hayan tenido éxito. Como se ha mencionado anteriormente, una misma cosa es
cierta para las bacterias que no tienen el gen cap FliD, como Shigella. Estas bacterias pueden tener todos
los otros genes flagelares, pero han perdido el gen cap —y no pueden producir
un flagelo ni han re-evolucionado el gen cap. ¿Por qué será?
Motorizar al
Flagelo
Ok, digamos que de alguna manera una colonia primitiva de
bacterias fue capaz de desarrollar algún proto-TTSS y un proto-filamento en donde
cada sistema fue independientemente funcional de alguna forma selectivamente
benefica. Ahí es en donde Matzke argumenta que sería cosa muy fácil juntar esos
dos sistemas para obtener la motilidad flagelar.
Antes de analizar esto, hagamos un poco de revisión.
Recuerde que el motor flagelar está dividido en dos unidades básicas —el
estator y el rotor. El estator se compone de las subunidades MotA y MotB (cada
una compuesta por aproximadamente 300 aminoácidos). El rotor se compone de FliM
(~330 aa), FliN (~130 aa), y la FliG (~330 aa). Estos 3 componentes del rotor también
están involucrados en la construcción del flagelo. El anillo-C, formado por esos
componentes, actúa como una especie de copa de medición que determina el tamaño
del gancho. Lo que ocurre es que aproximadamente 120 monómeros de gancho se
unen a FliM, FliN y FliG, (en 4 sitios
de unión en cada uno). Cuando se saturan todos los sitios de unión, todos los
monómeros se liberan a la vez y se forma un segmento de "gancho" de
una longitud específica. Después de que los monómeros de gancho abandonan el
anillo C, otra proteína entra y convierte el anillo en C de un secretor de
proteínas del gancho a un secretor de proteínas de flagelina. La especificidad del
anillo C cambia con respecto a qué tipo de monómeros se acepta.
De esta manera, la FliG es importante en la construcción
del flagelo en el sentido de que se precisa de sus 200 residuos N-terminales.
De hecho, si uno se divide la cadena de 331 aa de la FliG en segmentos de 10 aa
cada uno, mutaciones deletéreas en los segmentos 11, 13, 16, 17, 20, 21 y 27
dan como resultado la no-formación de flagelo adecuado y, obviamente, eso
afecta a la función de motilidad. También, aquellas bacterias con mutaciones en
los segmentos 1, 3, 12, 14, 15, 22, 23, y 26 de la FliG son completamente
"no-flageladas" [10].
Eso significa que el planteo de Matzke de que FliG, como
parte del complejo de proto-secreción, es "retenido sólo con el fin de
estabilizar/sostener al complejo secreción co-adaptado y al anillo FliF, y [es]
por lo contrario, vestigial " es un completo disparate. La FliG es vital
para la secreción y no tiene nada que ver con la estabilización de FliF (FliF
ha demostrado ser bastante estable de forma independiente). Es sólo que FliF
sin FliG no puede formar un flagelo adecuado.
Por supuesto, la proteína FliG (sin contrapartes
homólogas significativas por cierto) es también la sub-parte responsable de
convertir la fuerza proto-motriz en fuerza de torca para el movimiento de
rotación del flagelo. Los ~100 residuos C-terminales parecen ser necesarios
para esta función. Además, mutaciones específicas en los segmentos 10, 18, 19,
24, 25, 28, 29 y 31 forman flagelos, pero los tales están paralizados. [10].
En cuanto a función rotativa, FliM y FliN son
responsables de la conmutación —el movimiento de una dirección a la otra—, no
de la generación de fuerza de torsión. Sin embargo, FliM y FliN siguen siendo
necesarias para la construcción del flagelo.
Ahora, vamos a hablar de FliF (el anillo MS de ~ 550 aa.
que forma el complejo del poro de la membrana) por un momento. La proteína FliF
no tiene homólogos conocidos fuera del TTSS (que se cree que ha evolucionado
desde el sistema flagelar —no al revés). Incluso teniendo en cuenta su
existencia en una proto-forma, explicar cómo un proto-flagelo/filamento podría
atascarse a ella de una forma beneficiosa es todo un reto. El ensamblaje de los
filamentos incluso más simples es bastante complicado, como se describió
anteriormente. Diversas proteínas chaperonas están involucradas en llevar
monómeros específicos a su lugar justo en el momento adecuado y también de
plegarlos y unirlos. La construcción de un pilus aparentemente simple es
extremadamente compleja. La construcción de un flagelo hueco que se forma
añadiendo monómeros al extremo distal es extraordinariamente complicado.
Teniendo en cuenta estos hechos presentados hasta el
momento, tengo sólo unas pocas preguntas. Matzke sugiere que FliG no necesitaba
evolucionar con FliF como parte del aparato de exportación. ¿Cómo se explica esto
ya que FliG es requerida para la construcción del flagelo? Si FliG no
evolucionó con FliF, entonces ¿sería necesario que se uniera fuertemente a FliF
de una manera tal que supera las fuerzas rotación de la FliG, y que también lo
haga de una forma en que ayude en la construcción del flagelo? No sólo que la
FliG debe unirse a la FliF, sino que también tiene que tener especificidad para
cierto tipo de monómero de filamento. A lo que quiero llegar es que sin la
afinidad especifica de la FliG por la flagelina, el flagelo no se forma. Cuando
el flagelo se empieza a formar, el anillo MS (FliF) y el anillo C (FliG N-term
+ FliN + FliM) deben formarse en primer lugar o no formarán el flagelo. Eso
parece un algo difícil de explicar a través de mecanismos evolutivos.
¿Acaso todo el
proceso evolutivo sería más fácil si FliG ya estuviese unida a FliF? ¿Si FliG
originalmente evolucionara junto a FliF? Porque entonces ya tendríamos la
afinidad específica para el monómero de filamento y el filamento flagelar ya
podría ser producido ¿verdad? Pero entonces, ¿cómo es que motA/B se unirian a
FliG de una forma benéfica? Un buen número de residuos específicos tienen que
ser alineados a la perfección para que la fuerza protón-motriz de motA/B sea transferida a FliG como fuerza de torca.
En fin, de cualquier manera en que uno lo mira entiende
que es mucho lo que se necesita para que se vinculen FliF con FliG. ¿Qué tan
útil sería que la FliG tenga afinidad especifica por la flagelina si no
estuviera unida a la FliF primero? ¿De qué serviría que la FliG tenga afinidad
específica por el componente motA/B si
no estuviera anclada a motA/B primero? La adquisición de estas afinidades
implicará numerosas diferencias adicionales en la posición de los residuos
aminoacídicos a partir de las "proto" formas. Y lo más probable es
que estas diferencias requeridas no serían secuencialmente beneficiosas de manera
que la selección natural pueda guiar su puesta a punto.
Además, no esperemos que cierto grado de unión no-covalente
vaya a suceder entre FliG y FliF con sólo una o dos posiciones de residuos
correctas en lugar de las 46 posiciones de residuos bastante específicos que son
usados para unir FliG con FliF en los flagelos modernos. Para superar los
efectos de embate del movimiento browniano, el flagelo tiene que girar muy
rápidamente (~100-300 rotaciones/segundo durante 3-4 segundos). Esto significa
que una gran cantidad de inercia y fuerza de cizallamiento debe ser superada
para mantener conectada a FliG con FliF. Un número significativo de los 46
residuos de que intervienen en el anclaje ("de tipo cerrojo") tendría
que estar en su posición, todos al mismo tiempo, con el fin de superar estas
fuerzas de cizallamiento en cualquier grado seleccionable. De hecho, se sugiere
por experimentos de deleción que sólo el segmento N-terminal 4 de la FliG puede
sufrir cambios significativos sin que haya una pérdida completa de la motilidad.
Las mutaciones en los primeros 3 segmentos N-terminal (~30 aa.) dieron como
resultado una pérdida completa de la motilidad —obviamente debido a que no hay
la suficiente resistencia de unión a la FliF y/o una deficiencia de la capacidad
de contribuir en la formación del flagelo. [10]
Sin embargo, da la casualidad de que los genes para las
proteínas FliF y FliG están situados uno al lado del otro en el genoma. Ciertas
mutaciones de deleción entre FliG y FliF han resultado en una sola proteína
fusionada, una proteína FliG/FliF unida covalentemente que de hecho funciona
bastante bien. Es evidente que un enlace covalente es mucho más fuerte que una
unión no-covalente, por lo que se elimina la necesidad de tener decenas de uniones
no-covalentes colocadas en la posición adecuada. Aunque la proteína de fusión
unida covalentemente no funciona tan bien como el sistema de tipo silvestre que
no está unido covalentemente, funciona lo suficientemente bien como para
realizar el trabajo.
Debido a esta capacidad de la FliG de unirse de forma
covalente con FliF algunos me han dicho que esto hace que sea fácil de lograr
que los dos sub-sistemas (el motor y el rotor) se unan para dar lugar al super-sistema que cumple la función de la motilidad
flagelar. Esto no es así debido a la necesidad de la FliG de cumplir múltiples
roles en ambos sistemas al mismo tiempo —como se describe anteriormente.
Experimentos mutagenicos realizados en FliF muestran que
es necesario un "corto tramo C-terminal" de 9 residuos de aminoácidos
"esenciales" para que el proceso de "construcción del flagelo"
pueda darse. Tenga en cuenta que este proceso de montaje ocurre en el momento en
que el motor está apagado y no hay rotación de la FliG. Los autores afirman que:
"La eliminación o sustitución de hasta 10 aminoácidos inmediatamente por
encima de la región esencial dieron como resultado un flagelo paralizado."
[11] Eso suena como si fuese
bastante específica. Los autores dijeron que la eliminación o sustitución de 10
residuos adicionales resultaron en
parálisis flagelar. Parece, pues, que la rotación flagelar requiere algo
estructuralmente específico además de lo que se requiere para la construcción
flagelar. Un total de aproximadamente 19 aa bastante especificados de la
proteína FliF necesita estar en su lugar, tanto para la construcción del flagelo como para que la motilidad pueda hacerse
realidad.
Autor: Sean D. Pitman. Estudio en la Escuela de Medicina de Loma Linda University durante 1993-1997. Hizo su residencia en patología en el Centro Médico de Loma Linda University entre 2001-2005, y posteriormente trabajó en el área de hematología en City of Hope National Medical Center, durante 2005-2006. Tiene diversas publicaciones científicas.
Traductor: Daniel Alonso - Estudia Licenciatura en Ciencias Biológicas en la UNT , Argentina.
REFERENCIAS:
[1] Nicholas
Matzke, Evolution in (Brownian) space: a model for the origin of the bacterial
flagellum, talkreason.org, 2003 ( http://www.talkreason.org/articles/flagellum.cfm )
[2] Anand Sukhan, Tomoko Kubori, James Wilson, y Jorge
E. Galin. 2001. Genetic Analysis of Assembly of the Salmonella enterica
Serovar Typhimurium Type III Secretion-Associated Needle Complex. J. Bacteriology 183: 1159-1167.
[3] Macnab, R. M., 1999. The bacterial flagellum:
reversible rotary propellor and type III export apparatus. J Bacteriology. 181 (23), 7149-7153.
[4] He, S. Y., 1998. Type III protein secretion in plant and animal
pathogenic bacteria. Annual Reviews in Phytopathology. 36, 363-392.
[5] Kim, J. F., 2001. Revisiting the chlamydial
type III protein secretion system: clues to the origin of type III protein
secretion. Trends Genet. 17 (2), 65-69.
[6] Plano, G.
V., Day, J. B. and Ferracci, F., 2001. Type III export: new uses for an old
pathway. Mol Microbiol. 40 (2), 284-293.
[7] Nguyen,
L., Paulsen, I. T., Tchieu, J., Hueck, C. J. and Saier, M. H., Jr., 2000.
Phylogenetic analyses of the constituents of Type III protein secretion
systems. J Mol Microbiol Biotechnol. 2 (2), 125-144.
[8] Macnab, R. M., Science 290, p. 2087
[9] Macnab R. M., Bacteria create natural nanomachines,
USA Today, 2005
(http://www.USAtoday.com/weather/science/aaas/flagella121500.htm)
[10]May Kihara, Gabriele U. Miller, and Robert M.
Macnab, Deletion Analysis of the Flagellar Switch Protein FliG of Salmonella,
J. Bacteriol. 2000 June; 182(11): 3022-3028. (http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=94485)
[11] Bjorn Grunenfelder, Stefanie Gehrig, and Urs
Jenal,Role of the Cytoplasmic C Terminus of the FliF Motor Protein in Flagellar
Assembly and Rotation, Journal of Bacteriology, Mar. 2003, p. 1624-1633
Vol. 185, No. 5 (http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=148050&blobtype=pdf )
[12] Todas las
animaciones presentadas en este ensayo son fruto del esplendido trabajo de Keiichi
Namba et al. del ERATO Protonic NanoMachine Project (http://www.npn.jst.go.jp/index.html)
[13] Mis
agradecimientos a Mike Gene por la excelente información dada sobre el falgelo bacteriano en este
website: (http://www.idthink.net/)





No hay comentarios :
Publicar un comentario